Covid 19 - Casos y Examenes - Santiago de Chile
Actualmente hay mucha conversación sobre cuándo y cómo puede comenzar el desconfinamiento en Santiago de Chile. Como ya se mencionó en un post anterior sobre movilidad, cinco comunas en la conurbación de Santiago y dos más en la Región Metropolitana iniciaron la fase dos de el plan de desconfinación Paso a Paso.
Este plan tiene cinco pasos de desconfinamiento desde la cuarentena completa hasta la apertura avanzada. Hay varios criterios sobre cuándo una comuna puede avanzar o retroceder. Los criterios son:
- Ocupación UCI (cama de cuidados intensivos) Regional
- Ocupación UCI Nacional
- Tasa R comunal
- Tasa proyectada de casos regionales
- Positividad regional
- Porcentaje de casos aislados en 48 horas
- Porcentaje de casos nuevos que provienen de contactos de seguimiento
Teniendo en cuenta esta información, está claro que los datos sobre el número de casos son muy importantes. Si se analizan cada día los datos de nuevos casos conocidos en la Región Metropolitana se muestra que los casos han ido disminuyendo. Sin embargo, para comprobar si esta mejora es real, es necesario comparar el número de casos con la cantidad de exámenes de PCR. Este es el objetivo de este post.
2) Paquetes
Se utilizan los siguientes paquetes en esta publicación.
library(ggplot2)
library(dplyr)
library(stringr)
library(readr)
library(lubridate)3) Datos
Se utilizan datos del Ministerio de Salud de Chile, que se pueden descargar desde su github. Especificamente se utilizan los siguientes datos:
- producto 3 - casos accumulados de Covid 19 a nivel regional
- producto 7 - examenes PCR a nivel regional
4) Comportamiento de Casos Conocidos en Región Metropolitana
Los datos se trazan para casos en la Región Metropolitana para ver su comportamiento. Por lo tanto, primero se realiza alguna ingeniería de características para preparar los datos.
Se filtran los datos para incluir solo datos de la Región Metropolitana. Luego se calcula el número de casos nuevos cada día. Se modifica la cifra de nuevos casos del 17 de junio, ya que ese día se sumaron 31.412 casos, que antes se desconocían, al total de casos conocidos. Por tanto, para el 17 de junio hay una cifra de 32.230 casos nuevos en la Región Metropolitana. Esta cifra se cambia a 4.022 casos para que los datos solo reflejen específicamente nuevos casos conocidos cada día.
Metropolitana_Casos_Dia <- CasosTotalesCumulativo_T %>% select(Region, Metropolitana) %>%
mutate(NuevosCasosDia = Metropolitana - lag(Metropolitana))
Metropolitana_Casos_Dia[107,03] <- 4022
ggplot(data = Metropolitana_Casos_Dia) + geom_point(aes(x = Region, y = NuevosCasosDia)) + ylim(0,7000) + ggtitle("Nuevos Casos Región Metropolitana") + ylab("Nuevos Casos Conocidos") + xlab("Fecha")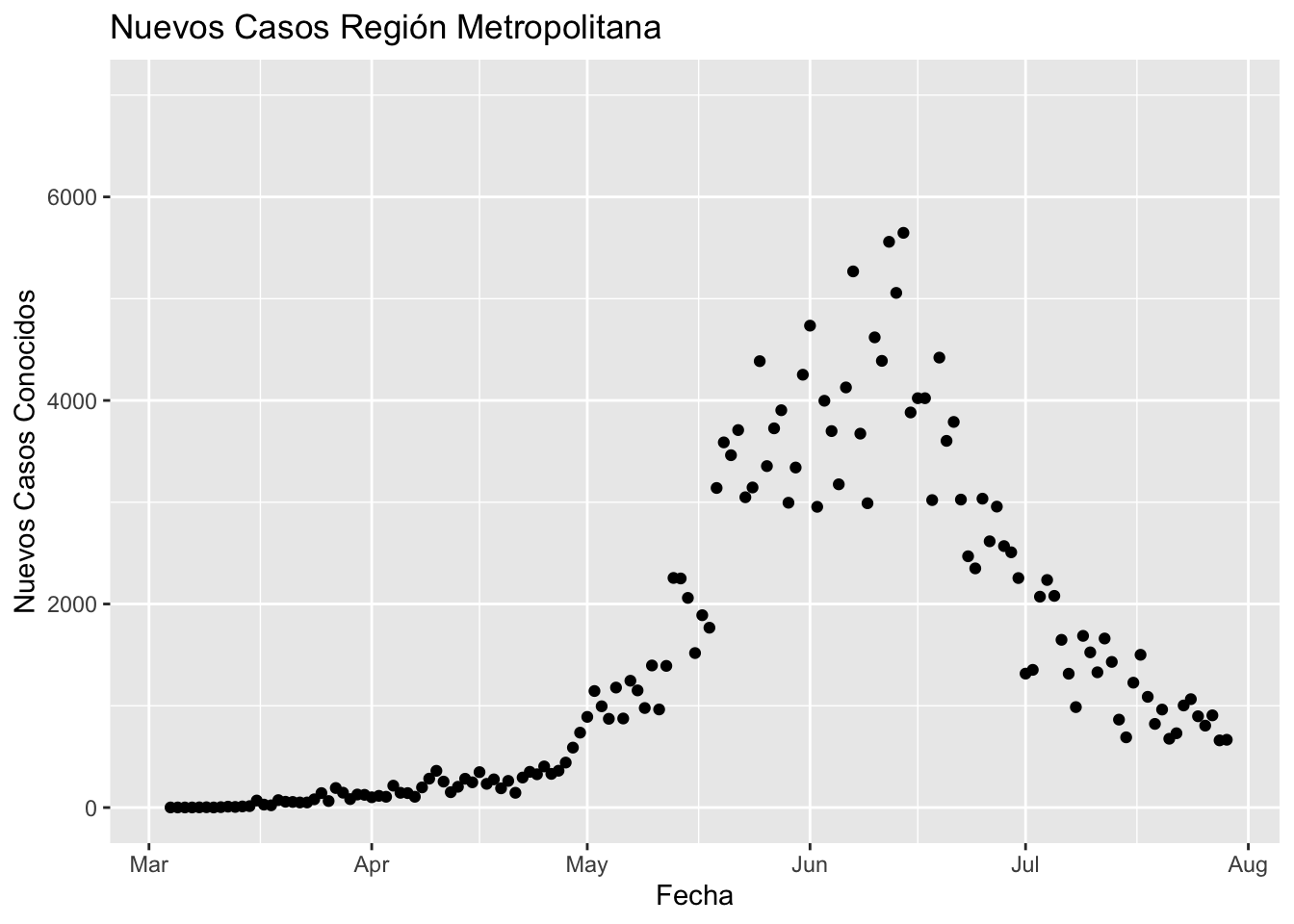
El gráfico anterior muestra que los casos nuevos en la Región Metropolitana aumentaron entre mayo y la primera quincena de junio. El punto más alto fue el 14 de junio con 5.647 nuevos casos registrados. Desde entonces, los casos han disminuido con 666 casos registrados hoy, 29 de julio. Es una buena noticia, pero ¿qué sucede cuando se comparan los nuevos casos con la cantidad de exámenes PCR?
5) Numero de Examenes PCR
Los datos sobre la cantidad de exámenes PCR diarias están disponibles a partir del 9 de abril. Por lo tanto, se crean nuevas observaciones para el 3 de marzo al 8 de abril, ya que el 3 de marzo es cuando comienzan los datos para nuevos casos.
Metropolitana_PCR <- PCR_T %>% select(Region, Metropolitana)
Metropolitana_PCR <- Metropolitana_PCR[-c(1:2),]
Metropolitana_PCR <- rbind(data_frame(Region = seq(as.Date("2020-03-03"), as.Date("2020-04-08"), "day"),
Metropolitana = rep(0, 37)), Metropolitana_PCR)Las dos bases de datos se combinan y se grafican los nuevos casos y los exámenes PCR.
Metropolitana_Casos_PCR <- left_join(Metropolitana_Casos_Dia, Metropolitana_PCR, by = "Region")
colnames(Metropolitana_Casos_PCR) <- c("Fecha", "CasosAcumulados", "NuevosCasosDía", "ExamenesPCR")
ggplot(data = Metropolitana_Casos_PCR) + geom_point(aes(x = Fecha, y = NuevosCasosDía, color = ExamenesPCR)) +
scale_color_continuous(low = "blue", high = "green") + ggtitle("Casos Conocidos y Examenes PCR en la Región Metropolitana") + ylab("Casos Nuevos") + xlab("Fecha")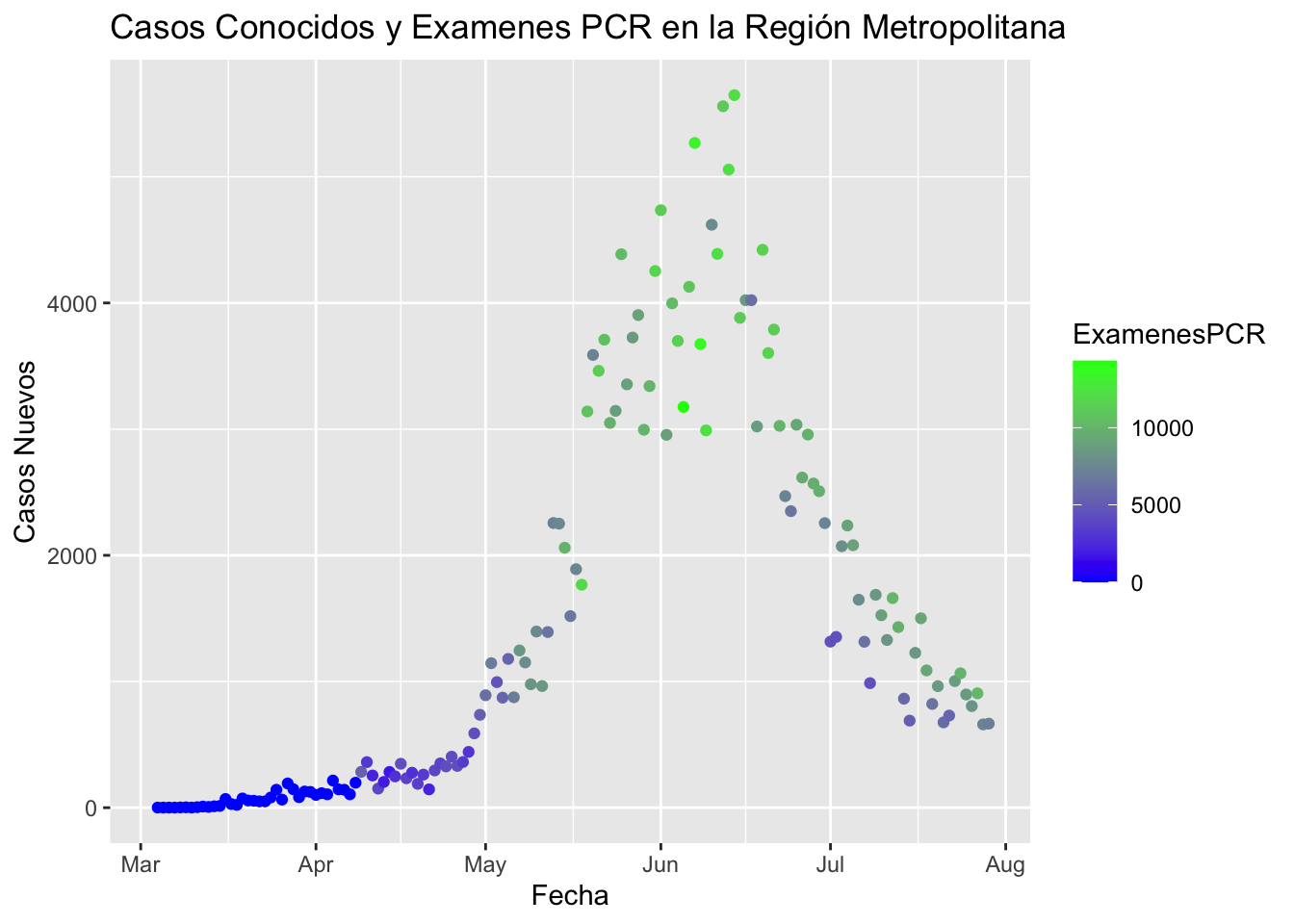
El gráfico anterior muestra cómo entre mediados de junio y finales de julio el número de nuevos casos ha disminuido, pero al mismo tiempo también el número de exámenes ha disminuido. Las siguientes tablas muestran el número promedio de examenes PCR diarias y casos nuevos diarios durante períodos de 15 días a partir de principios de mayo.
Metropolitana_Casos_PCR$Mes <- month(Metropolitana_Casos_PCR$Fecha)
Metropolitana_Casos_PCR$DiaMes <- day(Metropolitana_Casos_PCR$Fecha)1 - 15 de mayo
Promedio de exámenes PCR por dia = 7.171 Promedio de casos Nuevos por dia = 1.310
Metropolitana_Casos_PCR %>% filter(Mes == 5 & DiaMes %in% c(1:15)) %>% summary() #7171 #1310 ## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR Mes
## Min. :2020-05-01 Min. :10516 Min. : 872 Min. :4570 Min. :5
## 1st Qu.:2020-05-04 1st Qu.:14118 1st Qu.: 971 1st Qu.:6284 1st Qu.:5
## Median :2020-05-08 Median :17979 Median :1151 Median :7173 Median :5
## Mean :2020-05-08 Mean :18550 Mean :1310 Mean :7171 Mean :5
## 3rd Qu.:2020-05-11 3rd Qu.:22013 3rd Qu.:1394 3rd Qu.:8092 3rd Qu.:5
## Max. :2020-05-15 Max. :29276 Max. :2256 Max. :9948 Max. :5
## DiaMes
## Min. : 1.0
## 1st Qu.: 4.5
## Median : 8.0
## Mean : 8.0
## 3rd Qu.:11.5
## Max. :15.016 - 31 de mayo
Promedio de exámenes PCR por dia = 9.561 Promedio de casos Nuevos por dia = 3.202
Metropolitana_Casos_PCR %>% filter(Mes == 5 & DiaMes %in% c(16:31)) %>% summary() #9561 #3202## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-05-16 Min. :30794 Min. :1518 Min. : 6555
## 1st Qu.:2020-05-19 1st Qu.:40282 1st Qu.:3036 1st Qu.: 8680
## Median :2020-05-23 Median :52972 Median :3348 Median : 9853
## Mean :2020-05-23 Mean :53902 Mean :3202 Mean : 9561
## 3rd Qu.:2020-05-27 3rd Qu.:66987 3rd Qu.:3713 3rd Qu.:10658
## Max. :2020-05-31 Max. :80504 Max. :4386 Max. :11992
## Mes DiaMes
## Min. :5 Min. :16.00
## 1st Qu.:5 1st Qu.:19.75
## Median :5 Median :23.50
## Mean :5 Mean :23.50
## 3rd Qu.:5 3rd Qu.:27.25
## Max. :5 Max. :31.001 - 15 de junio
Promedio de exámenes PCR por dia = 11.555 Promedio de casos Nuevos por dia = 4.252
Metropolitana_Casos_PCR %>% filter(Mes == 6 & DiaMes %in% c(1:15)) %>% summary() #11555 #4252## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-06-01 Min. : 85239 Min. :2955 Min. : 7792
## 1st Qu.:2020-06-04 1st Qu.: 97478 1st Qu.:3686 1st Qu.:10992
## Median :2020-06-08 Median :112136 Median :4128 Median :11550
## Mean :2020-06-08 Mean :112833 Mean :4252 Mean :11555
## 3rd Qu.:2020-06-11 3rd Qu.:126914 3rd Qu.:4896 3rd Qu.:12278
## Max. :2020-06-15 Max. :144280 Max. :5647 Max. :14331
## Mes DiaMes
## Min. :6 Min. : 1.0
## 1st Qu.:6 1st Qu.: 4.5
## Median :6 Median : 8.0
## Mean :6 Mean : 8.0
## 3rd Qu.:6 3rd Qu.:11.5
## Max. :6 Max. :15.016 - 30 de junio
Promedio de exámenes PCR por dia = 9.144 Promedio de casos Nuevos por dia = 3.111
Metropolitana_Casos_PCR %>% filter(Mes == 6 & DiaMes %in% c(16:30)) %>% summary() #9144 #3111## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-06-16 Min. :148302 Min. :2255 Min. : 6143
## 1st Qu.:2020-06-19 1st Qu.:189776 1st Qu.:2538 1st Qu.: 7942
## Median :2020-06-23 Median :200861 Median :3021 Median : 9508
## Mean :2020-06-23 Mean :197809 Mean :3111 Mean : 9144
## 3rd Qu.:2020-06-26 3rd Qu.:210340 3rd Qu.:3696 3rd Qu.: 9918
## Max. :2020-06-30 Max. :219151 Max. :4421 Max. :11662
## Mes DiaMes
## Min. :6 Min. :16.0
## 1st Qu.:6 1st Qu.:19.5
## Median :6 Median :23.0
## Mean :6 Mean :23.0
## 3rd Qu.:6 3rd Qu.:26.5
## Max. :6 Max. :30.01 - 15 de julio
Promedio de exámenes PCR por dia = 7.321 Promedio de casos Nuevos por dia = 1.480
Metropolitana_Casos_PCR %>% filter(Mes == 7 & DiaMes %in% c(1:15)) %>% summary() #7321 #1480## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-07-01 Min. :220467 Min. : 690 Min. : 4439
## 1st Qu.:2020-07-04 1st Qu.:227168 1st Qu.:1316 1st Qu.: 5424
## Median :2020-07-08 Median :232158 Median :1431 Median : 7928
## Mean :2020-07-08 Mean :231984 Mean :1480 Mean : 7321
## 3rd Qu.:2020-07-11 3rd Qu.:237530 3rd Qu.:1674 3rd Qu.: 8963
## Max. :2020-07-15 Max. :241345 Max. :2236 Max. :10072
## Mes DiaMes
## Min. :7 Min. : 1.0
## 1st Qu.:7 1st Qu.: 4.5
## Median :7 Median : 8.0
## Mean :7 Mean : 8.0
## 3rd Qu.:7 3rd Qu.:11.5
## Max. :7 Max. :15.016 - 29 julio
Promedio de exámenes PCR por dia = 8.089 Promedio de casos Nuevos por dia = 929
Metropolitana_Casos_PCR %>% filter(Mes == 7 & DiaMes %in% c(16:29)) %>% summary() #8089 #929## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-07-16 Min. :242572 Min. : 660.0 Min. : 5588
## 1st Qu.:2020-07-19 1st Qu.:246224 1st Qu.: 748.8 1st Qu.: 7104
## Median :2020-07-22 Median :248854 Median : 901.5 Median : 8487
## Mean :2020-07-22 Mean :248928 Mean : 929.2 Mean : 8089
## 3rd Qu.:2020-07-25 3rd Qu.:251921 3rd Qu.:1049.5 3rd Qu.: 9078
## Max. :2020-07-29 Max. :254354 Max. :1501.0 Max. :10138
## Mes DiaMes
## Min. :7 Min. :16.00
## 1st Qu.:7 1st Qu.:19.25
## Median :7 Median :22.50
## Mean :7 Mean :22.50
## 3rd Qu.:7 3rd Qu.:25.75
## Max. :7 Max. :29.00Se crea un marco de datos llamado a para almacenar estas cifras y se grafican a continuación.
a <- tibble(Periodo = c("01 - 15 mayo", "16 - 31 mayo", "01 - 15 junio", "16 - 30 junio", "01 - 15 julio", "16 - 29 julio"),
NuevosCasosDiaPromedio = c(1310, 3202, 4252, 3111, 1480, 929), ExamenesPCRPromedio = c(7171, 9561, 11555, 9144, 7321, 8089))
a## # A tibble: 6 x 3
## Periodo NuevosCasosDiaPromedio ExamenesPCRPromedio
## <chr> <dbl> <dbl>
## 1 01 - 15 mayo 1310 7171
## 2 16 - 31 mayo 3202 9561
## 3 01 - 15 junio 4252 11555
## 4 16 - 30 junio 3111 9144
## 5 01 - 15 julio 1480 7321
## 6 16 - 29 julio 929 8089level_order_periodo <- c("01 - 15 mayo", "16 - 31 mayo", "01 - 15 junio", "16 - 30 junio", "01 - 15 julio", "16 - 29 julio")
ggplot(a) + geom_point(aes(x = factor(Periodo, levels = level_order_periodo) , y = NuevosCasosDiaPromedio), color = "red") +
geom_point(aes(x = factor(Periodo, levels = level_order_periodo), y = ExamenesPCRPromedio), color = "Blue") +
ggtitle("Promedio casos neuvos y examenes PCR por peridos de 15 días") + ylab("Nuevos Casos = Rojo / Examenes PCR = Azul") +
xlab("Periodo")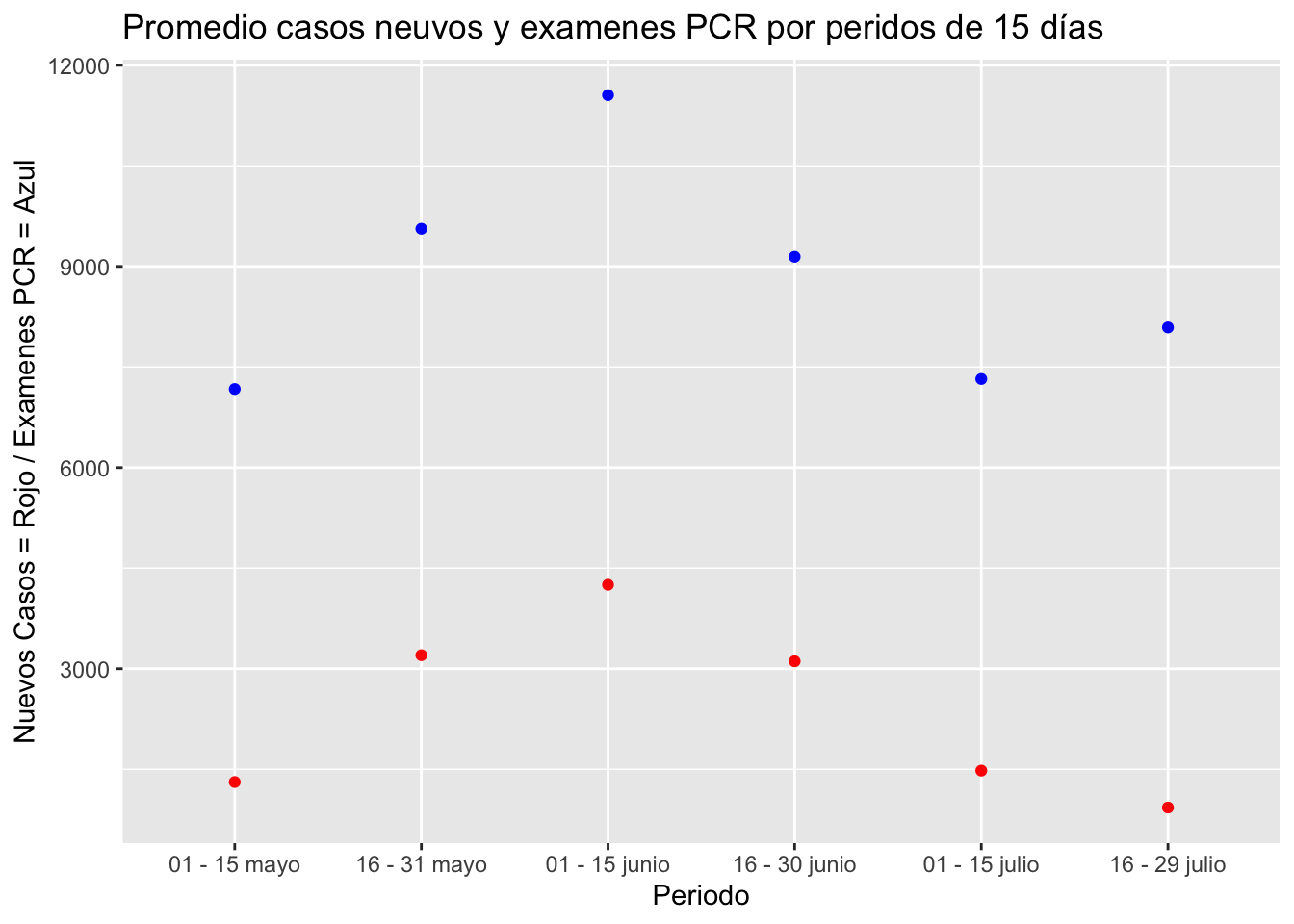
Este gráfico muestra varios puntos interesantes. Primero, muestra que la cantidad de casos nuevos y exámenes PCR en la Región Metropolitana han tenido comportamientos similares a lo largo de la pandemia. Cuando los casos han aumentado, los exámenes también han aumentado. Asimismo, cuando los casos han bajado, los exámenes también han bajado. Este ha sido el caso para todos los períodos excepto el más reciente (16 al 29 de julio). En la tabal a continuación se calculan las tasas de aumento y reducción para los casos y exámenes,y la tasa de positividad.
a %>% mutate(CasosRate = (NuevosCasosDiaPromedio / lag(NuevosCasosDiaPromedio))*100) %>% mutate(ExamenesRate = (ExamenesPCRPromedio / lag(ExamenesPCRPromedio))*100) %>% mutate(tasadepositividad = (NuevosCasosDiaPromedio/ExamenesPCRPromedio)*100) %>% select(Periodo, CasosRate, ExamenesRate, tasadepositividad)## # A tibble: 6 x 4
## Periodo CasosRate ExamenesRate tasadepositividad
## <chr> <dbl> <dbl> <dbl>
## 1 01 - 15 mayo NA NA 18.3
## 2 16 - 31 mayo 244. 133. 33.5
## 3 01 - 15 junio 133. 121. 36.8
## 4 16 - 30 junio 73.2 79.1 34.0
## 5 01 - 15 julio 47.6 80.1 20.2
## 6 16 - 29 julio 62.8 110. 11.5Las tasas de aumento y reducción de casos y examenes durante los períodos de estudio de 15 días muestran que:
Entre el 1 de mayo y el 15 de junio la tasa de aumento de los exámenes fue del 133% y el 121%. La tasa de aumento de casos fue positiva con 244% y 133%. Este comportamiento muestra que con más exámenes también aumenta el número de casos. También muestra que en ese período hubo una tasa de positividad muy alta. Por ejemplo, para el período del 16 al 31 de mayo, la tasa de positividad fue del 33,5%, es decir que el 33,5% de los exámenes para Covid 19 dieron positivo. Además, la tasa de positividad para el período del 1 al 15 de junio fue alta con un 36,8%.
A principios de julio el gobierno de Chile comenzó a hablar de una “leve mejoría” con una reducción de casos. Es cierto que los casos conocidos cayeron en la segunda quincena de junio, con un aumento de 73% (es decir una reduccion de 27%) respecto a la primera quincena del mes. Sin embargo, el número de examenes también descendió con una tasa de aumento muy similar de 79,1% (es decir una reduccion de 20,9%), además de que, la tasa de positividad se mantuvo muy alta en 34%.
En julio los casos han seguido bajando, y esta vez hay algunas noticias positivas. Primero, en la primera quincena de julio, los casos cayeron con un aumento de 47,6% (es decir una reduccion de 52,4%), mientras que los exámenes aumentaron un 80,1% (es decir una reduccion de 19,9%); lo que significa que para ese período la tasa de positividad fue del 20,2% y mucho más cercana al 15%, 10%, 5% y 1% requerido para pasar a diferentes pasos de desconfinamiento.
Además, en la segunda quincena de julio los casos han seguido disminuyendo, con un aumento en el número de exámenes. Esto ha resultado en una tasa de positividad del 11,5%.
Para resumir, estas cifras sugieren que la situación en Santiago está mejorando. En la segunda quincena de junio cuando disminuyeron los casos, fue solo un reflejo de la reducción de exámenes, pero desde entonces los casos han seguido disminuyendo, con la cantidad de exámenes PCR disminuyendo mucho menos o incluso aumentando.
El gráfico siguiente ilustra este punto con más detalle con la normalización de casos que se muestra, es decir, cuántos casos habría habido cada día si se hubieran realizado 9206 (tercer cuartil de exámenes por día) cada día.
summary(Metropolitana_Casos_PCR$ExamenesPCR)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0 1656 6547 5845 9206 14331Metropolitana_Casos_PCR <- Metropolitana_Casos_PCR %>% mutate(CasosNormalizados = (9206/ExamenesPCR)*NuevosCasosDía)
is.na(Metropolitana_Casos_PCR) <- sapply(Metropolitana_Casos_PCR, is.infinite)
Metropolitana_Casos_PCR[is.na(Metropolitana_Casos_PCR)] <- 0ggplot(data = Metropolitana_Casos_PCR) + geom_line(aes(x = Fecha, y = NuevosCasosDía), color = "red") +
geom_line(aes(x = Fecha, y = CasosNormalizados)) + ggtitle("Casos Conocidos vs. Casos Normalizados") + ylab("Rojo = Casos Conocidos, Negro = Casos Normalizados")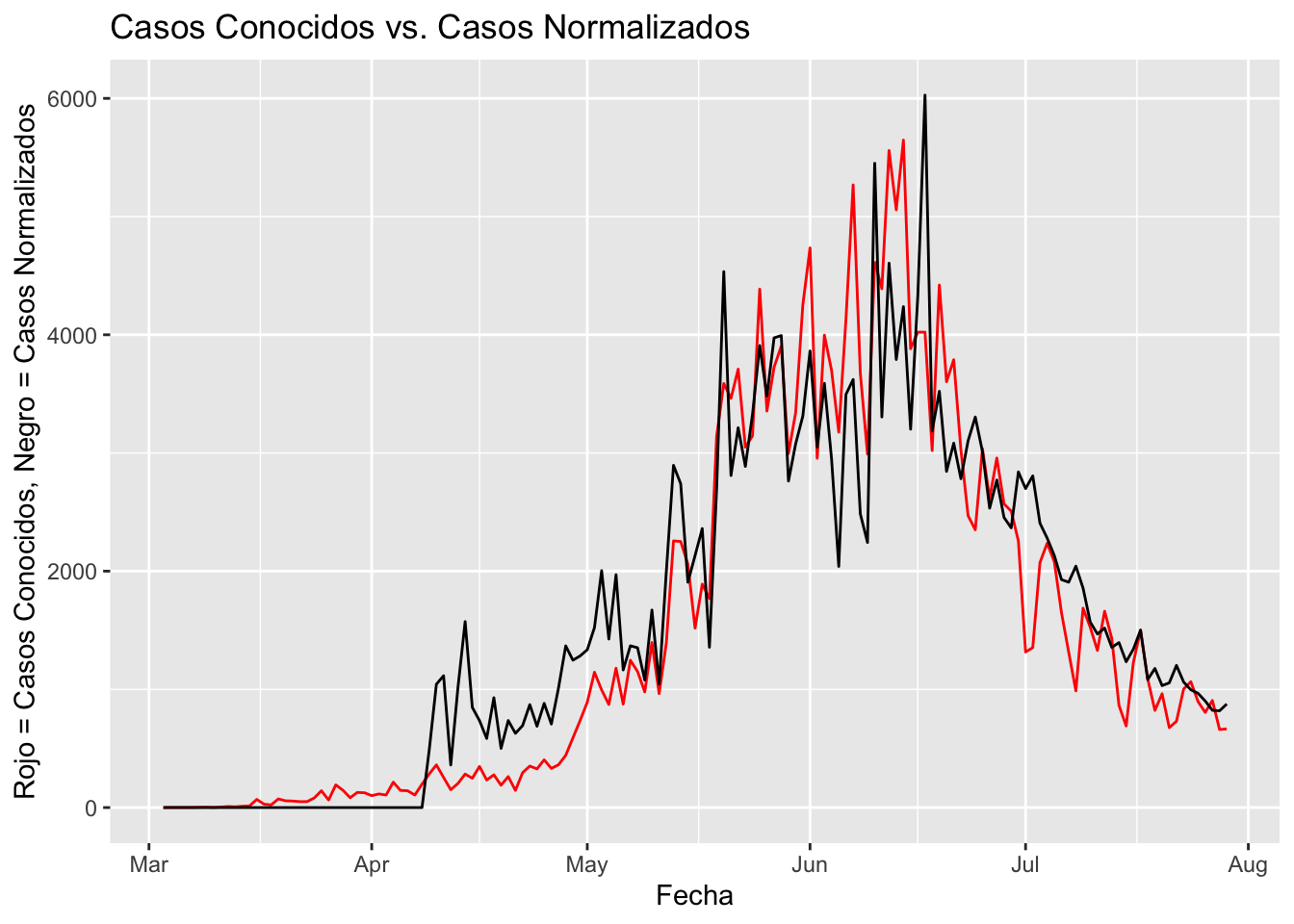
El gráfico muestra que el número de casos normalizados ha seguido el mismo patrón que los casos reales en julio. Sí, los casos normalizados son un poco más altos, con una diferencia promedio en julio de +284 casos, pero se están reduciendo. Por tanto, es justo decir que ha habido una “leve mejoría”. Sin embargo, la lucha contra el virus debe continuar en Chile, respetando las reglas del distanciamiento social, el uso de una mascarilla y también siempre sería bueno aumentar la cantidad de exámenes de PCR con exámenes hechos al azar. Con más exámenes, se comprenderá mejor la situación real con respecto a covid 19. Muchas gracias por leer esta publicación, y ojalá que haya sido informativa.
Metropolitana_Casos_PCR %>% filter(Mes == 7) %>% mutate(Diferencia = CasosNormalizados - NuevosCasosDía) %>% summary()## Fecha CasosAcumulados NuevosCasosDía ExamenesPCR
## Min. :2020-07-01 Min. :220467 Min. : 660 Min. : 4439
## 1st Qu.:2020-07-08 1st Qu.:232158 1st Qu.: 864 1st Qu.: 6349
## Median :2020-07-15 Median :241345 Median :1088 Median : 8332
## Mean :2020-07-15 Mean :240164 Mean :1214 Mean : 7692
## 3rd Qu.:2020-07-22 3rd Qu.:248352 3rd Qu.:1501 3rd Qu.: 8983
## Max. :2020-07-29 Max. :254354 Max. :2236 Max. :10138
## Mes DiaMes CasosNormalizados Diferencia
## Min. :7 Min. : 1 Min. : 818.8 Min. :-142.81
## 1st Qu.:7 1st Qu.: 8 1st Qu.:1055.1 1st Qu.: 44.85
## Median :7 Median :15 Median :1354.9 Median : 139.41
## Mean :7 Mean :15 Mean :1497.6 Mean : 283.74
## 3rd Qu.:7 3rd Qu.:22 3rd Qu.:1906.7 3rd Qu.: 379.15
## Max. :7 Max. :29 Max. :2806.0 Max. :1452.97